编辑|璐璐有话说
«——[·引言·]——»
SAR11细菌群是一类广泛存在于海洋中的细菌,其在碳循环和生物多样性维持中起着重要作用,本次研究中,通过对SAR11细菌群体的全基因组测序和比较分析,我们发现了高度多样化的SAR11分支中存在着简化和核心基因组保护的遗传策略。
实验结果表明,简化基因组是指通过减少基因数目和功能的方式,使细菌在特定环境中更加灵活和适应,我们观察到,在不同的海洋环境中,SAR11细菌具有类似的简化基因组,这可能是它们适应广泛环境的关键因素之一。
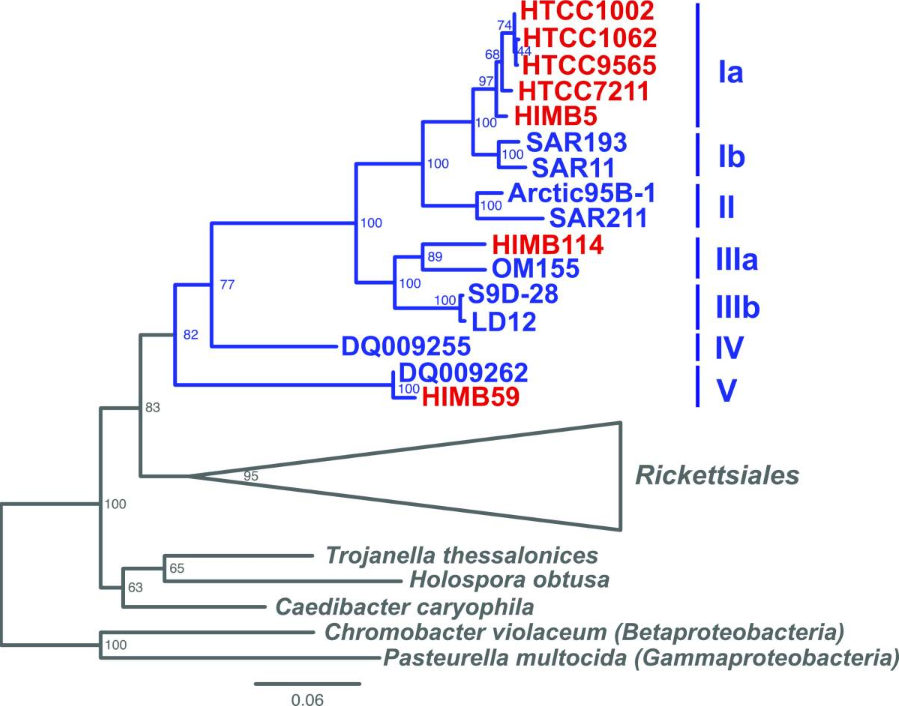
Ⅰ
«——[·SAR11亚支Ia基因组·]——»
基于与亚支Ia的其他基因组的共性,HTCC9565基因组的缺失信息量估计为1至约5.5kbp,虽然HIMB114基因组的完成程度更难估计,但最近测序的第二个亚支IIIa基因组的完成度为1.285Mbp,比目前的HIMB50序列大不到114kbp。
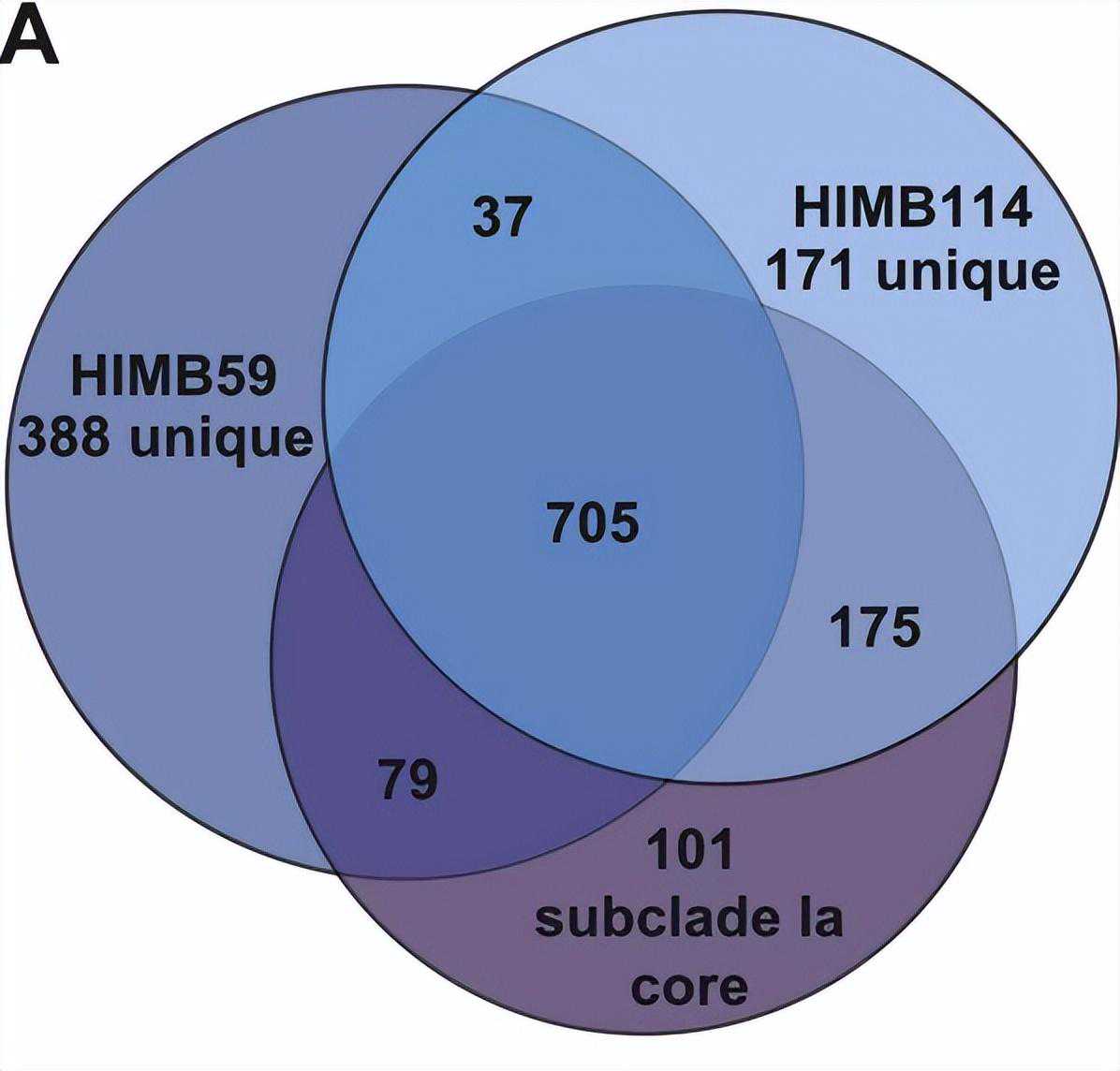
基因组编码1357至1576个基因,5S,16S和23S核糖体RNA基因的一个拷贝,以及30至35个tRNA,在任何菌株中均未发现假基因。
通过检查直系同源簇(OC)并排除副同源物和非蛋白质编码基因,研究了SAR11泛基因组,即在所有七个基因组中发现的基因总数,泛基因组总共包含2558个预测的OC,所有SAR705菌株中都存在11个OC的保守核心基因组。
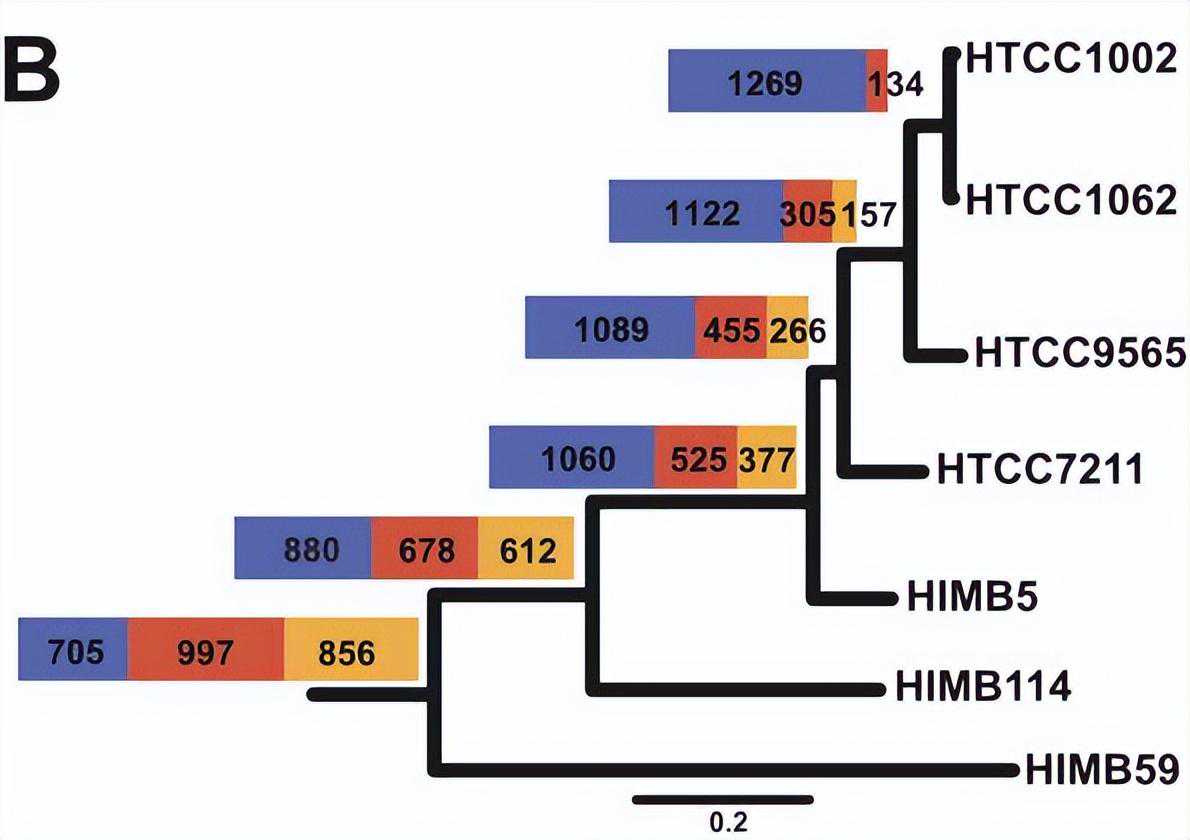
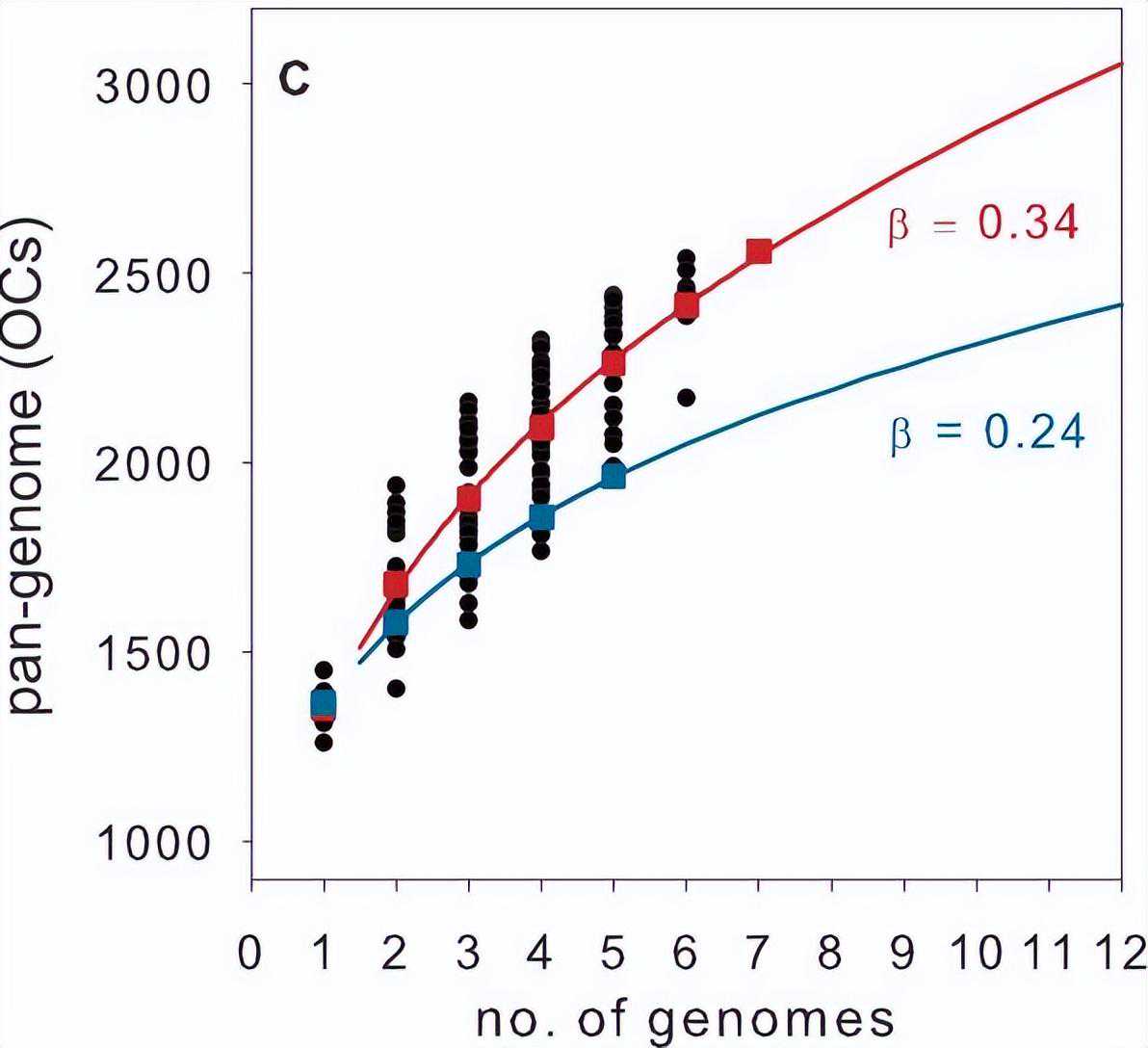
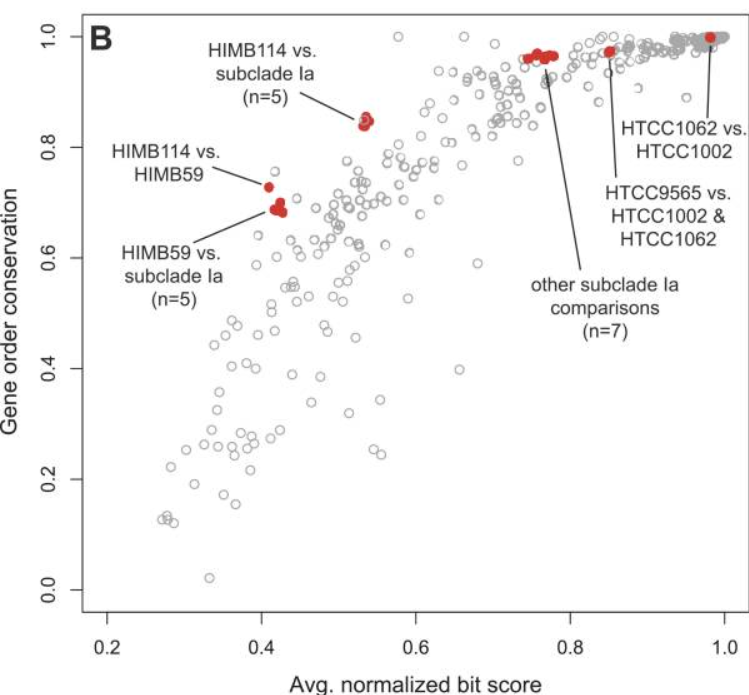
在不同系统发育相似性水平上,核心、唯一和共享的非核心OC对SAR11泛基因组的贡献发生了显着变化,核心基因组(蓝框)中的直系同源物数量与进化距离呈负相关。当仅考虑五个SAR11亚支Ia基因组时,1962个OC的泛基因组由1060个OC的更保守的核心基因组组成。
核心基因组和柔性基因组中基因的实际数量因系而异,核心SAR11和亚支Ia基因组的预测大小是通过将指数衰减函数拟合到为七个基因组序列的顺序相加计算的核心OC的平均数量。
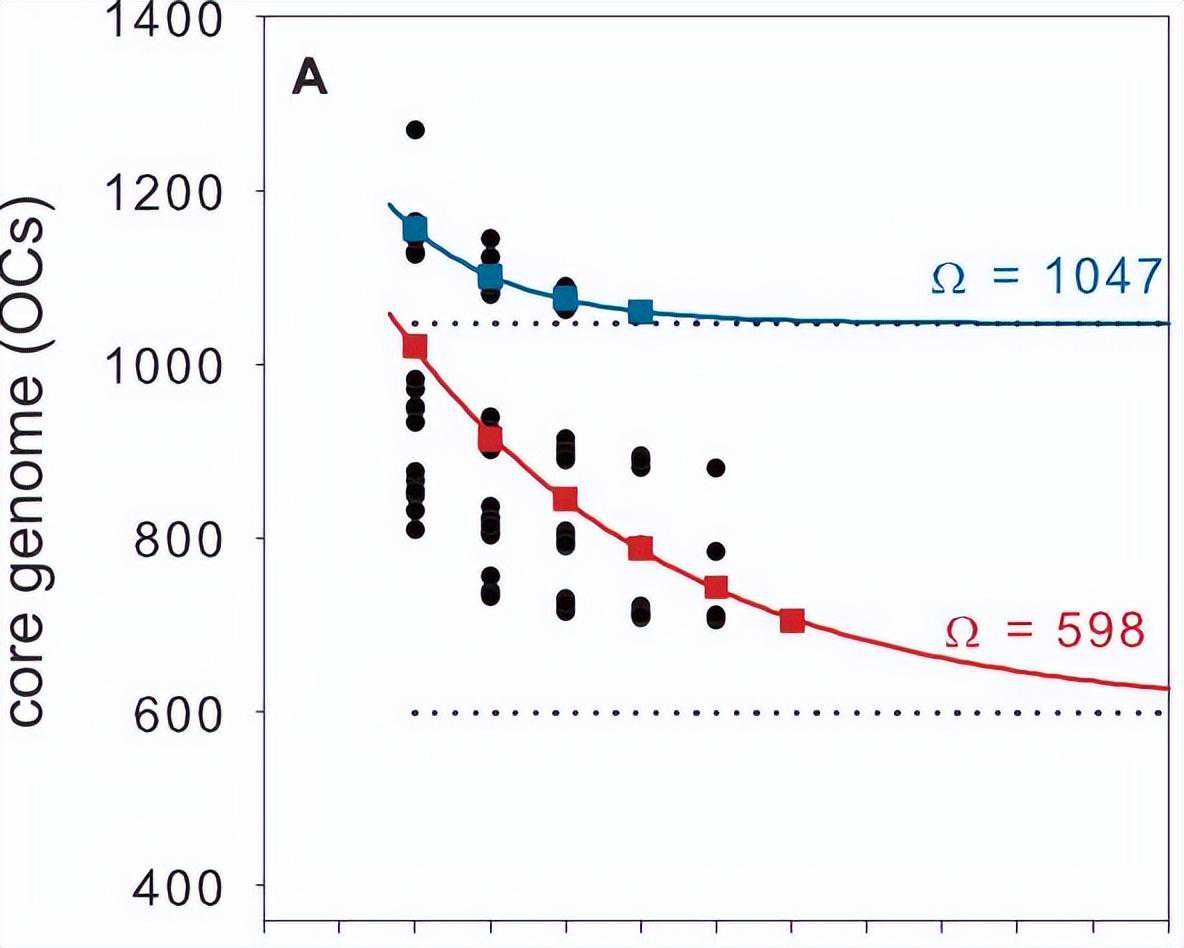
当分别考虑五个SAR11亚支Ia基因组时,预测的1047个OC的核心基因组与观察到的1060个核心基因组大小非常匹配,这表明当前的亚支Ia核心基因组是由可用基因组很好地定义的。
预测每个额外的测序基因组预期发现的新直系同源物的数量,以及SAR11泛基因组的开放程度,这意味着每个新测序的菌株将鉴定出多少个独特的基因。
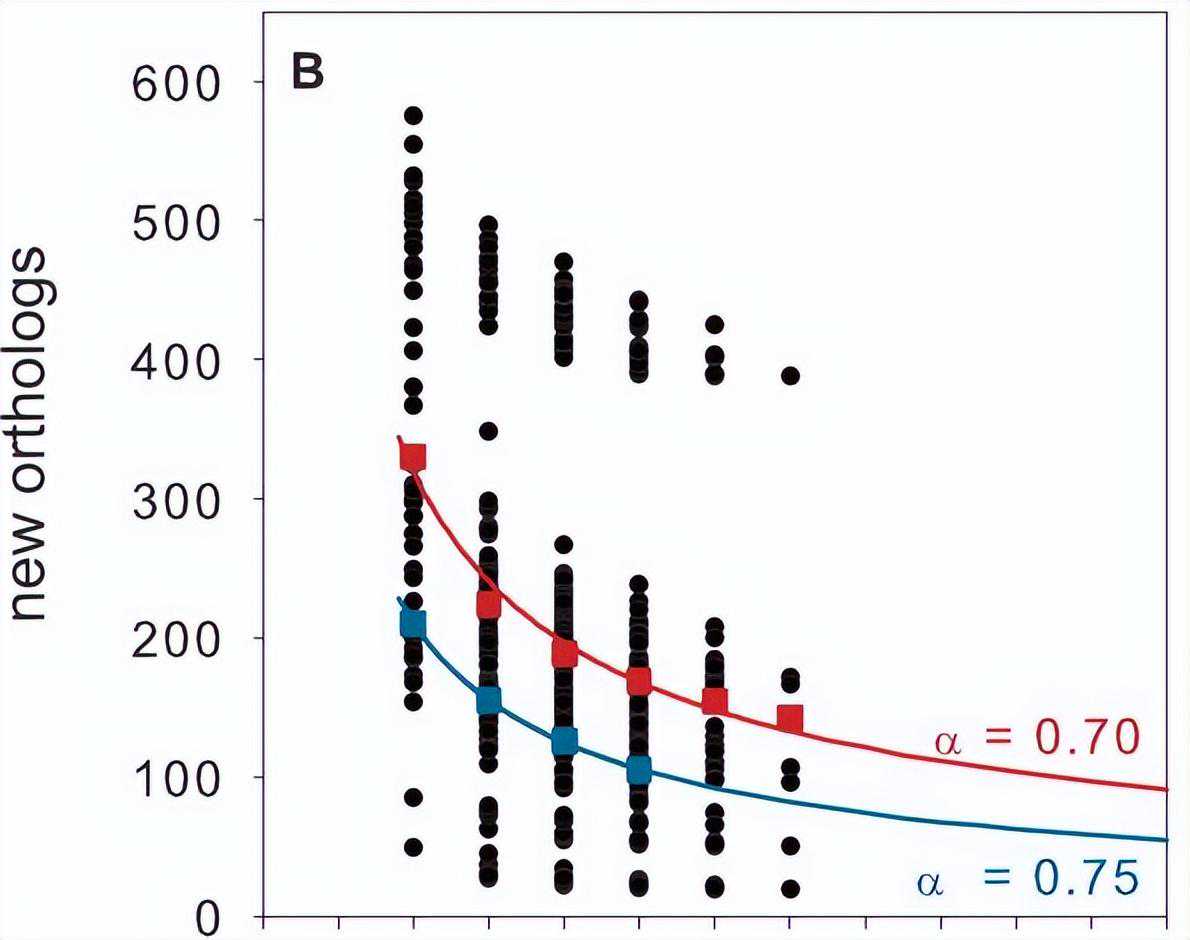
随着更多菌株的比较,新直系同源物的数量减少,当考虑所有七个测序基因组时,每个基因组的平均值为142个新直系同源物,当分别分析11个SAR5亚支Ia基因组时,第105个菌株增加的新直系同源物数量平均为11个。
对新SAR0直系同源物的平均数量和平均总泛基因组大小的幂律回归分析得出指数值为α=70.0和β=34,这些值与应用于泛基因组模型的堆定律所要求的关系,α≤1表示SAR11的开放泛基因组,虽然SAR11亚支Ia泛基因组也是开放的(α=0.75和β=0.24),但其较小的尺寸和较低的生长速率反映了该组比整个进化枝更好地被当前基因组定义。
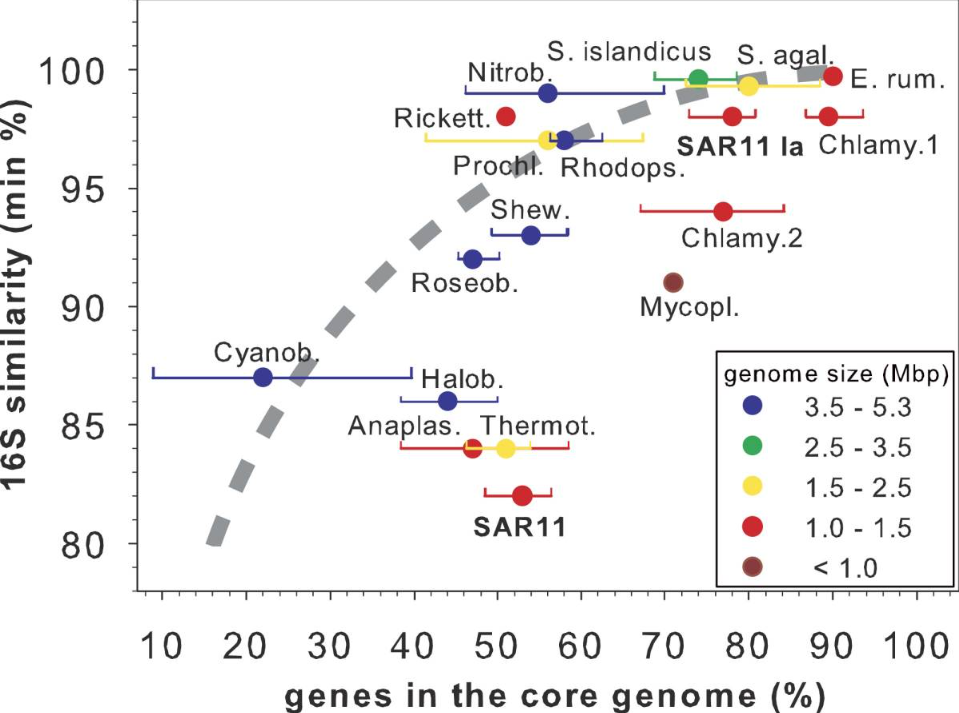
为了正确看待SAR11核心基因组的相对保守性,我们将我们的结果与其他比较基因组研究的结果进行了比较,包括对环境相关的原核生物和具有相似基因组大小,计算了核心基因的数量占每个SAR11菌株总基因的百分比。
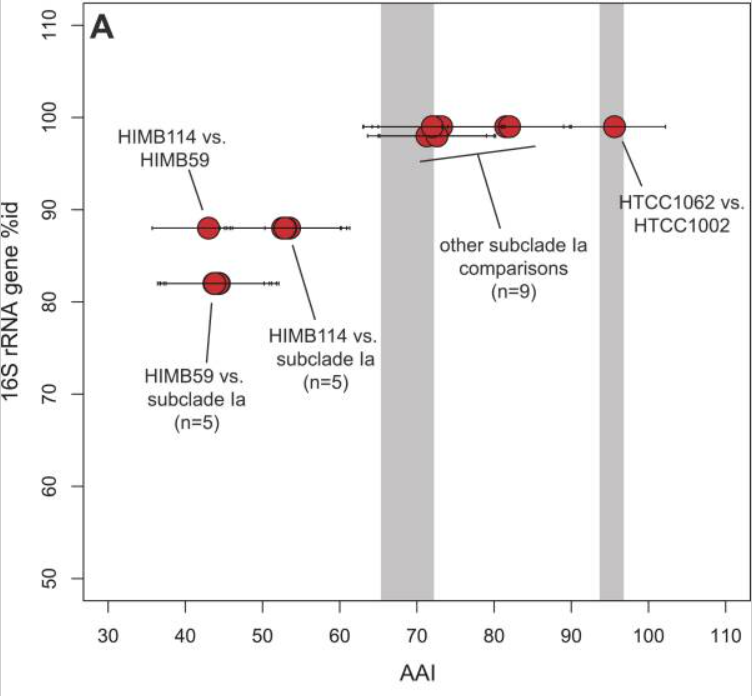
SAR11分支的成对平均氨基酸同一性(AAI)遵循细菌的一般趋势,并且基于16SrRNA基因比较(18%)和AAI比较,跨越阶级差异,SAR11核心基因组占每个菌株总基因库的48%至56%,在16SrRNA基因上具有相似分歧的组的核心基因组具有比SAR11核心基因组更小的平均保守性。
Ⅱ
«——[·SAR11核心基因组编码·]——»
具有16SrRNA基因分歧度的自由生活微生物的核心基因组与SAR11亚支Ia(2%)相似,例如原绿球菌(3%)或红假单胞菌(3%),其保守性要差得多,10个专性细胞内立克次体菌株的核心基因组在系统发育上与SAR11谱系密切相关,并且具有类似的小基因组大小(1.2Mbp),也比SAR11亚支Ia核心基因组保守得多。
基因组内基因顺序(synteny)的守恒可以成为保守基因功能和相关性的有力指标,先前的研究表明,同性随着系统发育距离而降低,尽管这种关系因所研究的组而异。
了可视化核心、附加亚支Ia核心、共享非核心和独特基因的全球基因组组织,我们对七个SAR11基因组进行了排序,方法是将它们共定位在dnaA,与HTCC1062的起源相邻,顺时针向dnaN移动。
与我们的计算显示共质高度保守一致,核心(和额外的亚支Ia核心)基因在整个基因组中分组,共享的非核心和独特基因分散在整个基因组中,一些密集分组的区域是显而易见的。
以16SrRNA,tRNA为界Ile-GAT,tRNA阿拉-TGC,一侧为23SrRNA盒,另一侧为5SrRNA,除HIMB59外,其HVR2由tRNA结合Ser-GGA和tRNAAla-GGC基因。
在HIMB59中,rRNA基因的顺序相同,但包括5SrRNA作为操纵子的一部分(16SrRNA,tRNAIle-GAT,tRNA阿拉-TGC,23SrRNA和5SrRNA),并且位于HVR2基因组的另一侧,在所有菌株中,HVR2区域在大小和位置上与dnaAN位点,包含~50个蛋白质编码基因,但HTCC7211和HIMB59除外,分别包含83个和74个基因。
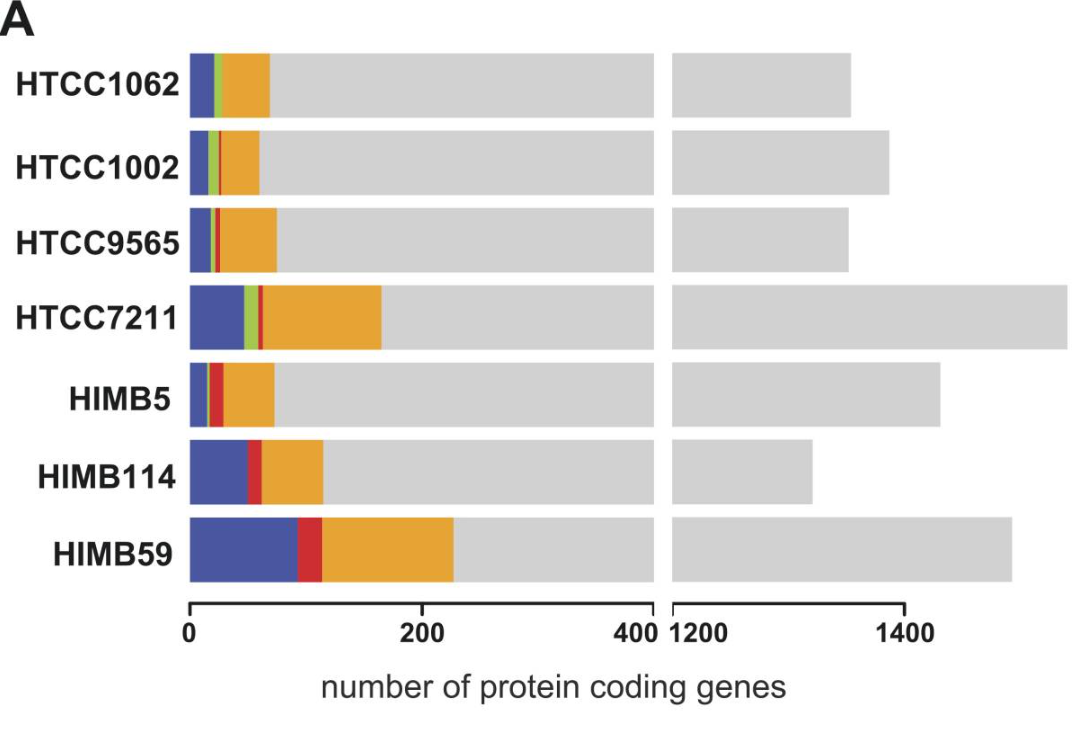
HVR2中常见的基因包括糖基转移酶,未知膜蛋白,假设蛋白和甲基转移酶,可能是因为HTCC1062和HTCC1002是最密切相关的菌株(AAI96%,ANI98%),它们共享HVR2中的所有基因。
剩余的分离株在该区域含有大量独特的基因,包括一些似乎赋予菌株特异性代谢能力的代谢能力,例如HTCC9565特有的硫代谢基因和HIMB59中的糖转运蛋白和磷酸果糖激酶,这可能表明该菌株的独特生态位。
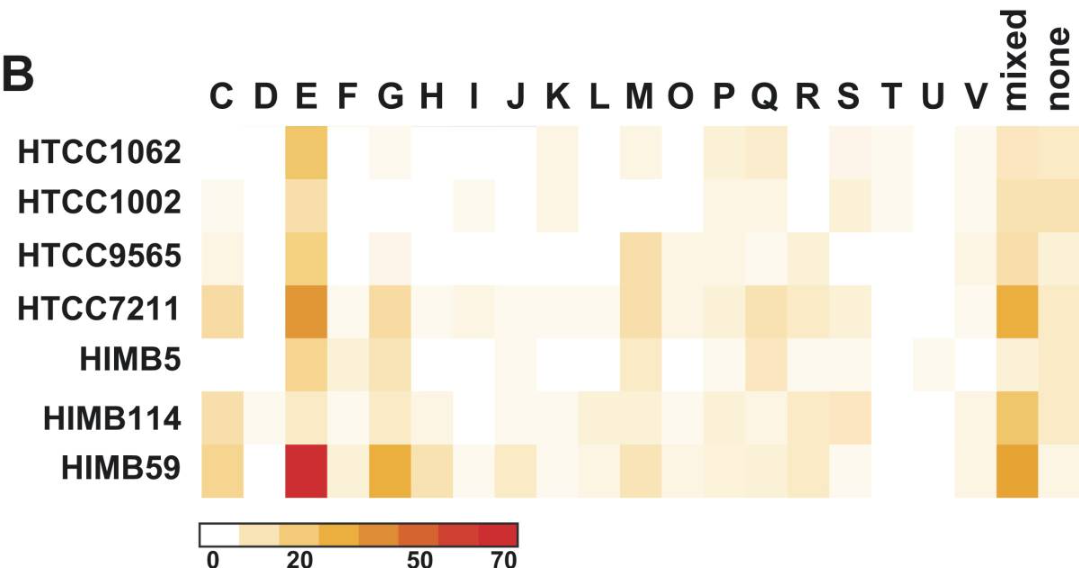
先前在HTCC1062的简化基因组中发现的一个显着特征是副同源物的发生率低,与这一发现以及细菌中基因组大小较小的副同源物数量减少的总体趋势一致,SAR11基因组范围为4.3%至15.2%的蛋白质编码基因作为副同源物,平均为7.8%。
菌株特异性副同源物的比例为每个基因组总副同源基因的18%至61%,泛基因组不同类别的基因分布相似,同源物和外同源物分别定义为给定物种形成事件之后或之前的重复。
我们用系统发育树表征了内同源物与外系同源物,确定相对于SAR11分支分化而发生的基因重复数量,在具有可靠系统发育分配的80%的副同源物中,19%被归类为外同源物。
大多数基因重复被预测发生在SAR11谱系与最后一个共同祖先分化之后,副同源物集中在几个COG(直系同源群簇)功能类别中。
能量产生和转化(C),氨基酸转运和代谢(E),碳水化合物运输和代谢(G),细胞壁/膜/包膜生物发生(M),以及具有混合名称,氨基酸转运和代谢(E)在七个SAR11基因组中占副同源物的数量最多,其中38%仅在HIMB59中发现。
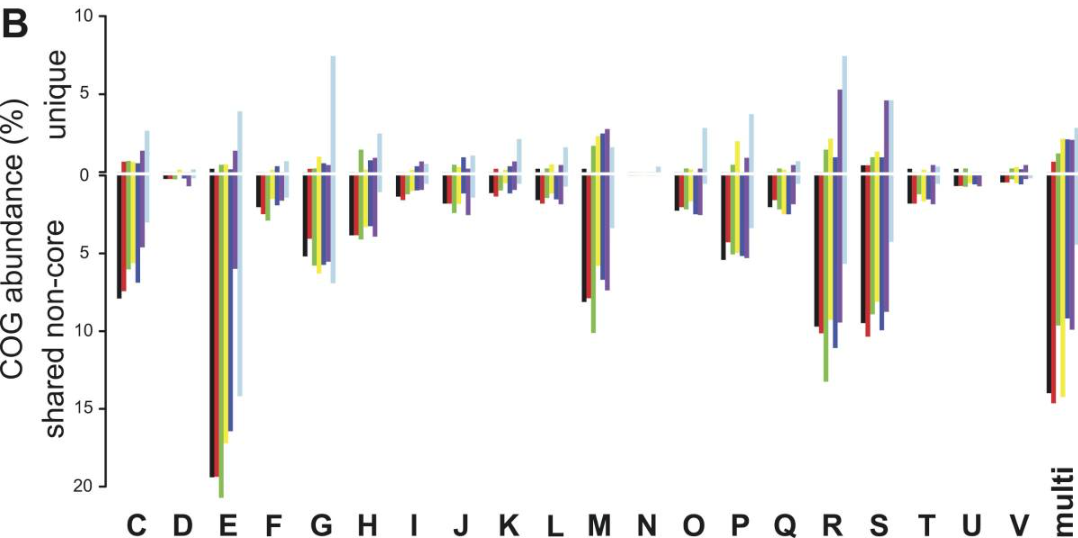
SAR11核心基因组具有高比例的编码参与管家功能和中枢代谢的蛋白质的基因,一小部分(2.1%至3.7%)的核心SAR11基因未被分配到COG功能类别。
.由于假定和假设基因的比例较大,柔性基因组中的未分类部分要高得多(23.5%至34.2%),与核心基因组相比,SAR11柔性基因组包括分配给COG类别氨基酸转运和代谢,碳水化合物转运和代谢。
所有SAR11基因组都编码蛋白视紫红质(PR),其存在于两组中,对不同的光波长具有特异性:绿色和蓝色吸收(分别为GPR和BPR)。
虽然已经发现GPR表层水域非常丰富,BPR主导着公海,如在马尾藻海,从开阔海域分离的两种SAR11菌株HTCC7211和HTCC9565含有BPR,而其余的沿海菌株则编码GPR,菌株HIMB114编码另一个发散的PR基因,具有目前未知的功能和吸收光谱。
从β-胡萝卜素,crtIBY和blh生物合成视网膜的假定基因存在于所有七个SAR11基因组中,本研究中检查的所有基因组中PR和相关基因的保守性表明,该基因在整个SAR11分支中具有重要的适应性作用,在碳限制期间,它通过提供ATP来潜在地促进生存。
«——[·结论·]——»
SAR11是一类在海洋中广泛分布的细菌,其基因组具有精简化的特点,这种基因组精简化可能是适应海洋生态系统的一种策略,使得SAR11细胞能够以更高的效率利用有限的资源,并在竞争中占据优势。
实验结果表明,SAR11细菌基因组中存在一个高变区域(HVR2),该区域的存在表明机械限制水平基因流不能解释SAR11基因组的小型化,高度一致性和基因保守性使得其基因组能够作为一个遗传资源,为适应性演化提供了可能。
探索简化和核心基因组保护策略,对SAR11细菌在不同环境中的生态适应性,可以更好地理解这一关键微生物群体在全球海洋生态系统中的作用。
参考文献:乔瓦诺尼,安德森,《海洋浮游细菌基因组的自然变异》
邓普斯,菲利克斯,《海洋浮游细菌分支的培养》
唐纳德,查尔斯,《海洋玫瑰杆菌的生态基因组学》



